بلات ( انگلیسی: Baʽalat Gebal ) یا همان «بانوی بیبلوس»، الهه شهر بیبلوس در فنیقیه در دوران باستان بود.

wiki: بلات
بلات (بیوانفورماتیک). بلات ( ابزار هم تراز سازی شبیه بلاست ) ( به انگلیسی: BLAT که مخفف واژگان BLAST - like alignment tool می باشد ) ، یک الگوریتم هم تراز سازی دو به دوی توالی است که در اوایل دهه ۲۰۰۰ میلادی توسط جیم کنت از دانشگاه سانتاکروز کالیفرنیا برای کمک به بازسازی و تفسیر ژنوم انسان ارائه شد. [ ۱]
این الگوریتم در ابتدا به منظور کاهش زمان لازم برای هم تراز سازی میلیون ها خوانش ژنوم موش با توالی ژنوم انسان طراحی شده بود. در آن زمان، ابزارهای هم تراز سازی، قادر به انجام این عملیات به نحوی که به روز رسانی بازسازی ژنوم را امکان پذیر سازد، نبودند. بلات در مقایسه با ابزارهای پیشین، در هم تراز سازی دی ان ای/ mRNA ( ار ان ای پیام رسان ) حدود ۵۰۰ برابر و در هم تراز سازی پروتئین/پروتئین حدود ۵۰ برابر سریع تر است. [ ۱]
بلات یکی از چندین الگوریتم ارائه شده برای آنالیز و مقایسه توالی های زیستی مانند دی ان ای ، ار ان ای و پروتئین می باشد که با هدف اولیه هم ساخت شناسی برای کشف کارکرد زیستی توالی های ژنومی طراحی شد. [ ۲]
این الگوریتم برخلاف الگوریتم های برنامه نویسی پویا از جمله الگوریتم نیدلمن - وانچ و اسمیت - واترمن تضمین نمی کند که که هم ترازی بهینه ( از نظر ریاضی ) را بین دو توالی بیابد. در عوض ابتدا سعی می کند که توالی های کوتاه که با احتمال بیشتری هم ساخت هستند را به سرعت شناسایی کند و سپس آن ها را هم تراز کرده و نواحی هم ساخت را گسترش دهد. [ ۳]
از بلات می توان برای هم تراز سازی توالی های دی ان ای، پروتئین و همچنین توالی های ترجمه شده نوکلئوتید ( mRNa و دی ان ای ) استفاده کرد ( این الگوریتم روی توالی های با مشابهت بسیار عمل می کند ) .
لازم است ذکر شود که جستجوی دی ان ای بیشتر در نخستی سانان و جستجوی پروتئین بیشتر در مهره داران کارامد است. [ ۱] [ ۴]
علاوه بر این، توالی های پروتئین نسبت به توالی های دی ان ای، برای تشخیص تطابق های با فاصله زیاد و هم چنین آنالیز میان گونه ای کار آمد تر هستند. [ ۵]
کاربردهای رایج بلات شامل موارد زیر می باشد:
• هم تراز سازی چندین توالی mRNA با یک بازسازی ژنوم برای پی بردن به مختصات ژنومی. [ ۶]
• هم تراز سازی یک توالی پروتئین یا mRNA از یک گونه بر پایگاه داده ای از توالی های یک گونه دیگر برای تعیین هم ساختی.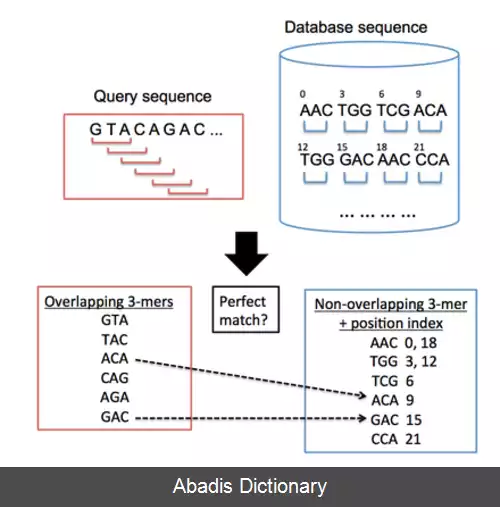
این نوشته برگرفته از سایت ویکی پدیا می باشد، اگر نادرست یا توهین آمیز است، لطفا گزارش دهید: گزارش تخلفاین الگوریتم در ابتدا به منظور کاهش زمان لازم برای هم تراز سازی میلیون ها خوانش ژنوم موش با توالی ژنوم انسان طراحی شده بود. در آن زمان، ابزارهای هم تراز سازی، قادر به انجام این عملیات به نحوی که به روز رسانی بازسازی ژنوم را امکان پذیر سازد، نبودند. بلات در مقایسه با ابزارهای پیشین، در هم تراز سازی دی ان ای/ mRNA ( ار ان ای پیام رسان ) حدود ۵۰۰ برابر و در هم تراز سازی پروتئین/پروتئین حدود ۵۰ برابر سریع تر است. [ ۱]
بلات یکی از چندین الگوریتم ارائه شده برای آنالیز و مقایسه توالی های زیستی مانند دی ان ای ، ار ان ای و پروتئین می باشد که با هدف اولیه هم ساخت شناسی برای کشف کارکرد زیستی توالی های ژنومی طراحی شد. [ ۲]
این الگوریتم برخلاف الگوریتم های برنامه نویسی پویا از جمله الگوریتم نیدلمن - وانچ و اسمیت - واترمن تضمین نمی کند که که هم ترازی بهینه ( از نظر ریاضی ) را بین دو توالی بیابد. در عوض ابتدا سعی می کند که توالی های کوتاه که با احتمال بیشتری هم ساخت هستند را به سرعت شناسایی کند و سپس آن ها را هم تراز کرده و نواحی هم ساخت را گسترش دهد. [ ۳]
از بلات می توان برای هم تراز سازی توالی های دی ان ای، پروتئین و همچنین توالی های ترجمه شده نوکلئوتید ( mRNa و دی ان ای ) استفاده کرد ( این الگوریتم روی توالی های با مشابهت بسیار عمل می کند ) .
لازم است ذکر شود که جستجوی دی ان ای بیشتر در نخستی سانان و جستجوی پروتئین بیشتر در مهره داران کارامد است. [ ۱] [ ۴]
علاوه بر این، توالی های پروتئین نسبت به توالی های دی ان ای، برای تشخیص تطابق های با فاصله زیاد و هم چنین آنالیز میان گونه ای کار آمد تر هستند. [ ۵]
کاربردهای رایج بلات شامل موارد زیر می باشد:
• هم تراز سازی چندین توالی mRNA با یک بازسازی ژنوم برای پی بردن به مختصات ژنومی. [ ۶]
• هم تراز سازی یک توالی پروتئین یا mRNA از یک گونه بر پایگاه داده ای از توالی های یک گونه دیگر برای تعیین هم ساختی.

wiki: بلات (بیوانفورماتیک)
