در ساختمان پروتئین، استراید ( ( STRIDE ( Structural identification ) یک الگوریتم برای انتساب ساختار ثانویه پروتئین از روی مختصات اتمی پروتئین بر پایهٔ به کارگیری ترکیبی انرژی پیوند هیدروژنی و اطلاعات آماری زاویهٔ پیچشی زنجیرهٔ اصلی است.
برای این که تعریف شهودی مارپیچ آلفا و صفحهٔ بتا ( که به طور معمول ساختار تعیین شده توسط بلورنگارها نمایانگر آن است ) تا حد ممکن دقیق تخمین زده شود، سهم پیوندهای هیدروژنی تشکیل دهندهٔ ساختار ثانویه و زوایای پیچشی زنجیرهٔ اصلی به صورت وزن دار در نظر گرفته می شود.
میزان مناسب بودن هر واحد مقدماتی ساختار ثانویه یا همان الگو ( پیچ های ۴ماندهای برای مارپیچ های آلفا و پل ها برای صفحات بتا ) به وسیله ترکیبی وزن دار از چند کمیت بیان می شود که هر کدام حاصل ضرب انرژی پیوندهای هیدروژنی مربوطه و تمایل مانده های اسید آمینه با زوایای φ و ϕ داده شده به حضور در مارپیچ های آلفا و صفحات بتا از لحاظ آماری است، و برای پذیرفتن یا نپذیرفتن یک الگو بر اساس میزان مناسب بودن آن، یک آستانه در نظر گرفته می شود. انتخاب تنها یک آستانه آن هم برای ترکیب کمیت های هر الگوی دارای پیوند هیدروژنی به جای در نظر گرفتن چند آستانه ( برای ترکیب های مختلف کمیت ها ) این اجازه را می دهد تا پارامترهای تشخیص با دقت بیشتری تعیین شوند؛ چرا که الگوهای دارای زوایای پیچشی نامناسب، در صورتی که پیوندهای هیدروژنی قوی داشته باشند قابل قبول هستند و برعکس، پیوندهای هیدروژنی نسبتاً ضعیف با زوایای صحیح زنجیرهٔ اصلی قابل جبران شدن هستند.
انرژی پیوند هیدروژنی ( E hb ) از روی تابع تجربی انرژی که از بررسی حجم زیادی از داده های آزمایشی روی هندسه پیوندهای هیدروژنی در ساختارهای بلوری پلی پپتیدها، پپتیدها، اسیدهای آمینه، و ترکیب های آلی کوچک به دست آمده محاسبه می شود:
E hb = E r × E t × E p
که در آن E r میزان بستگی پیوند هیدروژنی به فاصله است، و E t و E p حاوی ویژگی های جهتی آن هستند. عبارت مربوط به فاصله، یک تابع ۶ - ۸ است:
E r = C r 8 + D r 6
که در آن C = − 3 E m r m 8 kcal Å 8 / mol ، D = − 5 E m r m 6 kcal Å 6 / mol ، r فاصلهٔ بین اتم دهنده و پذیرنده است، و E m و r m به ترتیب انرژی و فاصلهٔ بهینهٔ پیوند هیدروژنی هستند. برای پیوندهای هیدروژنی زنجیرهٔ اصلی که به صورت N − H ⋯ O هستند، E m = − 2. 8 kcal / mol و r m = 3. 0 Å . عبارات زاویه ای E t و E p به صورت زیر هستند: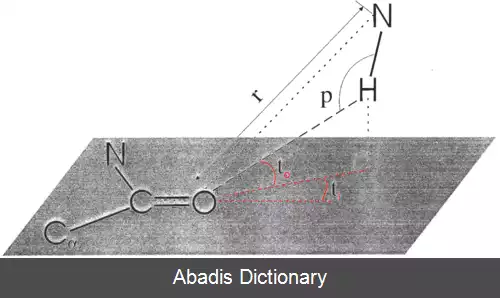
این نوشته برگرفته از سایت ویکی پدیا می باشد، اگر نادرست یا توهین آمیز است، لطفا گزارش دهید: گزارش تخلفبرای این که تعریف شهودی مارپیچ آلفا و صفحهٔ بتا ( که به طور معمول ساختار تعیین شده توسط بلورنگارها نمایانگر آن است ) تا حد ممکن دقیق تخمین زده شود، سهم پیوندهای هیدروژنی تشکیل دهندهٔ ساختار ثانویه و زوایای پیچشی زنجیرهٔ اصلی به صورت وزن دار در نظر گرفته می شود.
میزان مناسب بودن هر واحد مقدماتی ساختار ثانویه یا همان الگو ( پیچ های ۴ماندهای برای مارپیچ های آلفا و پل ها برای صفحات بتا ) به وسیله ترکیبی وزن دار از چند کمیت بیان می شود که هر کدام حاصل ضرب انرژی پیوندهای هیدروژنی مربوطه و تمایل مانده های اسید آمینه با زوایای φ و ϕ داده شده به حضور در مارپیچ های آلفا و صفحات بتا از لحاظ آماری است، و برای پذیرفتن یا نپذیرفتن یک الگو بر اساس میزان مناسب بودن آن، یک آستانه در نظر گرفته می شود. انتخاب تنها یک آستانه آن هم برای ترکیب کمیت های هر الگوی دارای پیوند هیدروژنی به جای در نظر گرفتن چند آستانه ( برای ترکیب های مختلف کمیت ها ) این اجازه را می دهد تا پارامترهای تشخیص با دقت بیشتری تعیین شوند؛ چرا که الگوهای دارای زوایای پیچشی نامناسب، در صورتی که پیوندهای هیدروژنی قوی داشته باشند قابل قبول هستند و برعکس، پیوندهای هیدروژنی نسبتاً ضعیف با زوایای صحیح زنجیرهٔ اصلی قابل جبران شدن هستند.
انرژی پیوند هیدروژنی ( E hb ) از روی تابع تجربی انرژی که از بررسی حجم زیادی از داده های آزمایشی روی هندسه پیوندهای هیدروژنی در ساختارهای بلوری پلی پپتیدها، پپتیدها، اسیدهای آمینه، و ترکیب های آلی کوچک به دست آمده محاسبه می شود:
E hb = E r × E t × E p
که در آن E r میزان بستگی پیوند هیدروژنی به فاصله است، و E t و E p حاوی ویژگی های جهتی آن هستند. عبارت مربوط به فاصله، یک تابع ۶ - ۸ است:
E r = C r 8 + D r 6
که در آن C = − 3 E m r m 8 kcal Å 8 / mol ، D = − 5 E m r m 6 kcal Å 6 / mol ، r فاصلهٔ بین اتم دهنده و پذیرنده است، و E m و r m به ترتیب انرژی و فاصلهٔ بهینهٔ پیوند هیدروژنی هستند. برای پیوندهای هیدروژنی زنجیرهٔ اصلی که به صورت N − H ⋯ O هستند، E m = − 2. 8 kcal / mol و r m = 3. 0 Å . عبارات زاویه ای E t و E p به صورت زیر هستند:

wiki: استراید
